BGISEQ PCR-free WGS 为您还原最真实的全基因组图谱
要点
避免PCR错误
最大程度还原基因组原始序列信息
避免PCR扩增偏向性
出色覆盖高GC/AT区域、启动子和重复序列等PCR建库方法无法覆盖的区域[1]
提高InDel检测的精准度和敏感度
高质量InDel从75%提升到86%,而低质量InDel从12%降低到3%[2]
适合肿瘤基因组研究
减少GC-bias,更全面的检测体细胞突变 [3][4]
高性价比
建库+测序100G,仅需4500元
无Index hopping担忧
BGISEQ平台PCR-free文库与常规PCR文库类似,污染率平均约为0.0004%[5]
从建库到测序的真正PCR-free
PCR-free建库 + DNB (DNA纳米球)核心测序技术[6],全方位实现PCR-free
简介
BGISEQ PCR-free WGS(人全基因组重测序)在文库构建过程中未进行PCR(聚合酶链式反应)扩增,从而避免了PCR错误和扩增偏向性的产生。且BGISEQ测序仪利用独特的DNA纳米球(DNB)技术,基于滚环复制(RCR)进行文库扩增,这种线性扩增可以避免常规PCR带来的错误累积,为您还原最真实的全基因组序列。
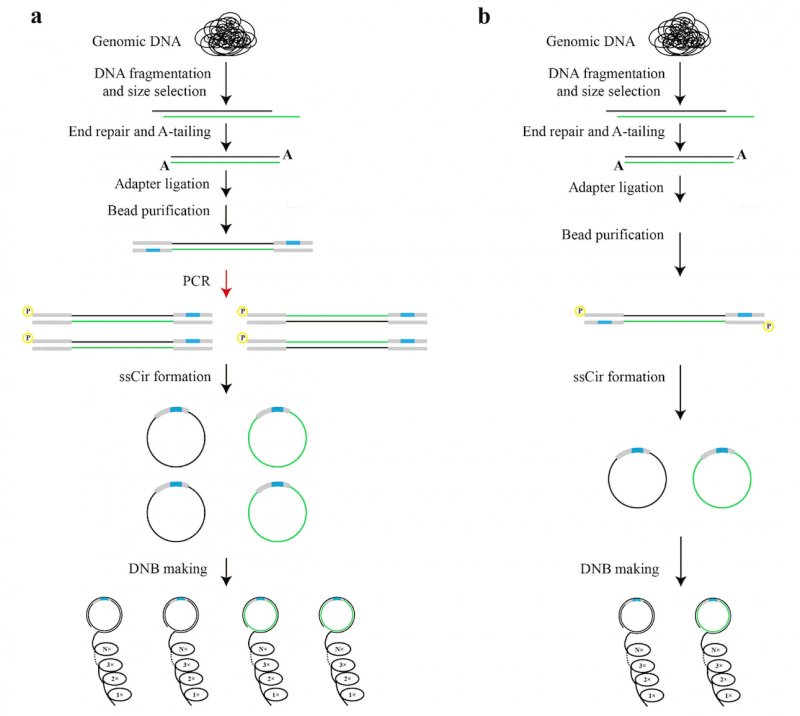
图1 BGISEQ PCR WGS(a)和BGISEQ PCR-free WGS(b)建库流程
送样建议
|
样本类型 |
总量 |
浓度 |
完整性(胶图) |
纯度 |
|
Genomic DNA |
≥1.5μg |
≥19ng/μL |
主峰>20Kb |
无蛋白,RNA/盐离子等污染,样本无色透明不粘稠 |
数据表现
1、标准品NA12878测序结果显示不同平台的PCR和PCR-free WGS数据的测序质量相当
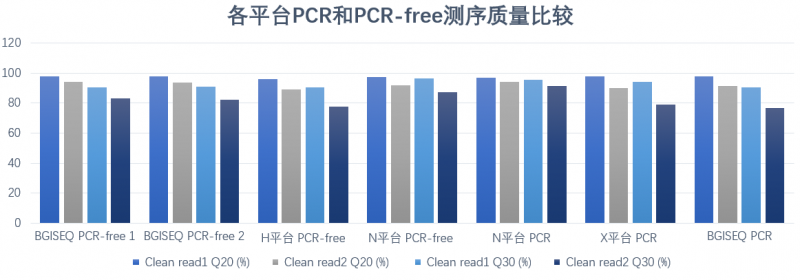
图2 各平台PCR和PCR-free测序质量比较
2、BGISEQ duplicate rate非常低(1.22%-1.95%),非BGISEQ平台即便是PCR-free建库,duplicates仍然较高(10%左右)
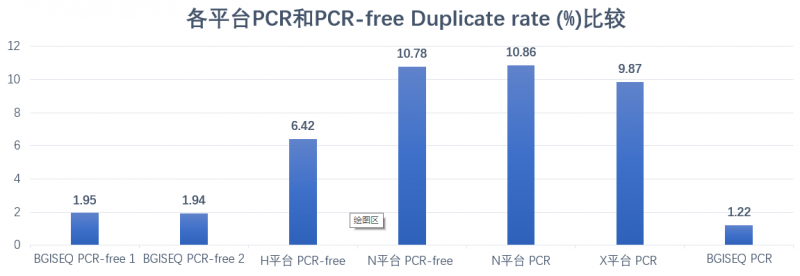
图3 各平台PCR和PCR-free duplicate rate比较
3、在不同平台上,PCR-free WGS InDel精准度和敏感度可高达99%和95%,均比PCR WGS 高出5%,PCR-free建库方法可明显提高InDel calling的准确性
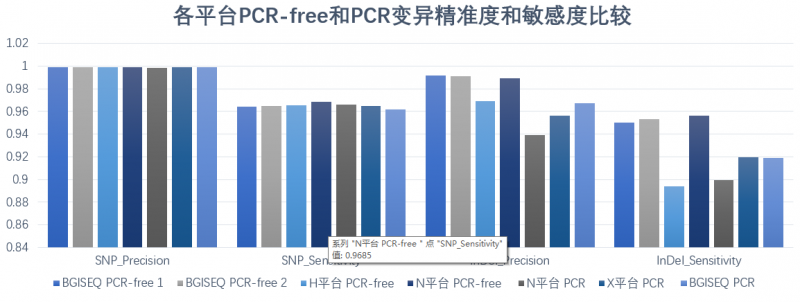
图4 各平台PCR-free和PCR变异精准度和敏感度比较
4、BGISEQ PCR-free相对于不同测序平台、不同建库方法,GC均一性表现最好
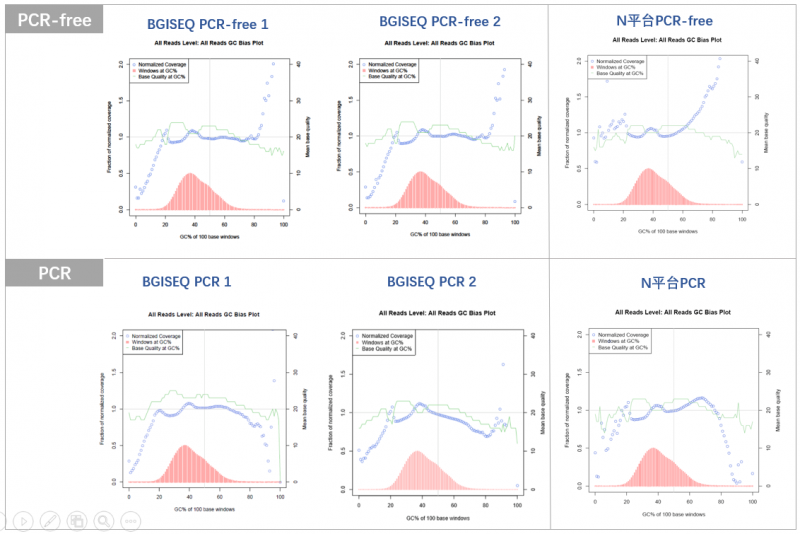
图5 各平台PCR-free和PCR GC均一性比较
(以100个碱基的大小为窗口,绘制GC含量分布图,结果越接近中间1.0的横线,表明覆盖深度越均一)
参考文献
【1】Kozarewa I, Ning Z, Quail M A, et al. Amplification-free Illumina sequencing-library preparation facilitates improved mapping and assembly of (G+C)-biased genomes[J]. Nature Methods, 2009, 6(4):291-295.
【2】Han F, Wu Y, Narzisi G, et al. Reducing INDEL calling errors in whole genome and exome sequencing data[J]. Genome Medicine,6,10(2014-10-28), 2014, 6(10):89.
【3】Nakagawa H, Wardell C P, Furuta M, et al. Cancer whole-genome sequencing: present and future[J]. Oncogene, 2015, 34(49):5943-50.
【4】Craig D W, Nasser S, Corbett R, et al. A somatic reference standard for cancer genome sequencing[J]. Scientific Reports, 2016, 6:24607.
【5】Li Q, Zhao X, Zhang W, et al. Reliable Multiplex Sequencing with Rare Index Mis-Assignment on DNB-Based NGS Platform. bioRxiv, 2018: 343137
【6】Drmanac R, Sparks A B, Callow M J, et al. Human genome sequencing using unchained base reads on self-assembling DNA nanoarrays.[J]. Science, 2010, 327(5961):78-81.

