- 首页 > 代谢组+16S关联分析
代谢组+16S关联分析
微生物组的研究已经逐渐进入到多组学的时代,越来越多的科学家将代谢组与微生物组关联起来,同时从微生物与代谢物两个角度分析生物学问题,通过解析微生物-代谢物的关联性,多维的角度揭示生命活动。
针对16S 及代谢组数据,华大基因基于代谢物丰度与微生物相对丰度进行联分析, 采取数据降维和相关性分析方法,分析两组学的整体相关性,探究生物学问题与代谢物和微生物之间的关系,找到高度相关的代谢物和微生物,进而挖掘微生物、代谢物以及所研究的生物学背景间存在的内在关联网络。
技术优势
1、关联分析研究经验丰富,在Nature等顶级期刊上自主发表多篇文献。
2、充分调研微生物组与代谢组关联分析文献,自主开发软件分析流程。
产品应用
1、 肠道微生物与疾病研究
2、 脑-肠-轴研究
3、 益生菌等微生物制剂开发
4、 根际微生物研究
5、 水体、土壤等环境研究
6、 茶叶、腐乳等发酵食品发酵工艺探索
技术路线
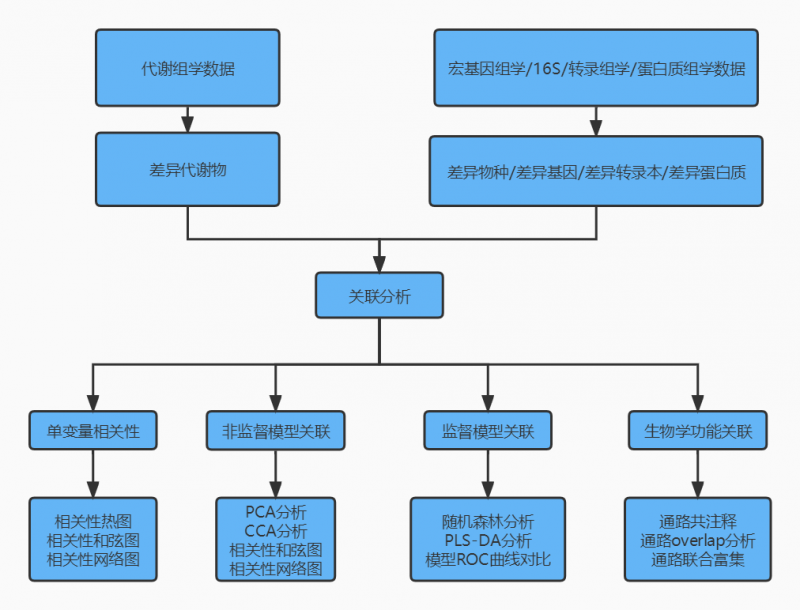
注:差异物种无生物学功能关联
项目周期
12个自然日
案例一:微生物与代谢关联揭示拟南芥三萜化合物对植物根系微生物组的调控规律[1]
实验设计:
选取野生型及突变型的模式物种拟南芥做根部代谢组分析及根部微生物群落16S分析,探究三萜化合物对植物根部微生物群落的调控作用。
主要结论:
16S分析显示拟南芥三萜化合物合成突变株的根微生物组成和多样性发生了变化,显示Bacteroidetes富集与Delatproteobacteria消耗的微生物调节模式。将其根部微生物与分类上较远物种水稻和小麦的根系细菌分布相比较,发现这些三萜化合物的生物合成途径能影响拟南芥根部微生物组的组成。代谢组分析发现拟南芥野生型及THAS过表达植株的根系中存在7种thalianol衍生产物(T1,T2,T9,T10,T18a-T18c)及arabidin(A5)在野生型型植株中不存在。
在三种主要的细菌门(Proteobacteria,Actinobacteria和Firmicutes)中选择了19种菌株分离纯化与三萜混合物培养,大多数Proteobacteria菌株增殖更快,而所有Actinobacteria菌株都被抑制,显示植物根部与土壤中的浓度和消耗模式一致,表明所试验的化合物有助于植物根际细菌的主动选择。对代谢敏感细菌属进行三萜化合物的体外培养实验,发现三萜代谢物可以选择性地调节这些根系细菌的生长。
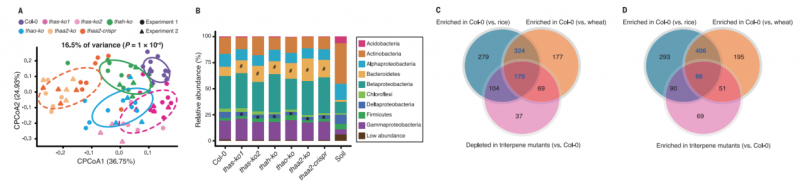
图1 三萜通路的突变体特异调控的根系细菌类群
参考文献
A specialized metabolic network selectively modulates Arabidopsis root microbiota. Science 2019
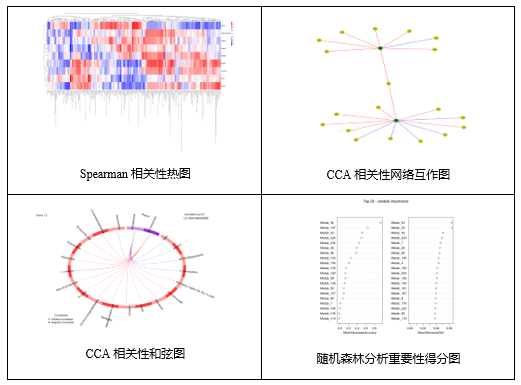
在关联分析中,综合运用了单变量相关性、非监督模型关联、监督模型关联、生物学功能关联。以下为部分分析结果图片展示:
1、 适用数据类型
要求用于关联分析的两组学的样本数据一一对应(即用于关联分析的两组学的样本来自于相同的生物学重复个体),代谢组数据可以为非靶向代谢组、脂质组学、靶向代谢组学中的任意一种。
2、 重复次数要求
1)当样本为2组时,每组样本数不少于8个;当样本组不少于3组时,每组样本不少于4个;每组样本大于10个时分析效果较好。
2)用于关联分析的临床样本推荐每组样本数不少于30个。
3)重复数少于建议数时分析效果差;每组低于3个样本时无法分析。
Q1:为什么用于关联分析的两组学样本要求一一对应?
A1:分析中包含Spearman相关性分析,其相关系数表明X(独立变量)和Y(依赖变量)的相关方向,在进行计算两组学的相关性时要求每一个组学数据要有能够对应的另一个组学数据,否则无法进行相关性分析。
Q2:16S+代谢关联分析对用于采集数据的样本有限制吗?
A2:没有限制。此关联分析针对16S及代谢组数据,满足条件的数据即可进行相应的关联分析,根据解读关联分析的结果进行数据挖掘,因此要求在前期设计实验时就要考虑到后面用于关联分析的两组学数据的样本来源在生物学上的相关性(一般建议用于关联分析的两组学数据来源于相同的生物学重复的个体),单纯的数据分析本身只讨论统计学的相关性。
Q3:对于关联分析的两组学数据的重复数的要求有什么依据吗?
A3:关联分析是依据16S及代谢组数据基础上进行的统计关联分析,故用于关联分析的样本数据越多,分析结果表现越好。
建议数是我们的项目经验值,当重复数低于建议数时,数据分析的表现结果差;且每组低于3个样本时无法进行关联分析。

