- 首页 > 单管单细胞 RNA-Seq
单管单细胞 RNA-Seq
单细胞RNA测序技术概述
目前高通量测序技术已经深入到人类疾病、物种进化、动植物分子育种等传统的生物学研究领域中,逐渐成为一种不可或缺的研究工具。然而随着生命科学和医学基础研究的深入发展,人们发现越来越多的特殊标本和特定的生物学现象,如法医鉴定的微量标本、肿瘤内部异质性等无法用常规组织测序的方法进行研究。单细胞测序技术的出现,给这类样本的研究带来了极大的便利。
单细胞测序技术经过了10余年的发展,取得了众多的技术进展与突破。其中,单细胞RNA测序技术中,Smart-Seq方法以其可实现完整mRNA扩增的优势,被广泛的应用。而近期广受关注的另外一个高通量单细胞RNA测序技术由Macosko E团队开发,其研究成果于2015年发表在《Cell》杂志上,该技术结合了微流控液滴法,磁珠对细胞进行标记,可一次性完成多至一百万个细胞分离和扩增建库,这个技术的出现大幅的降低了单细胞RNA测序的成本,使得单细胞RNA水平的研究得到了飞跃发展。
华大基因依托于这两个技术开发优化的相应单细胞RNA测序产品:单管单细胞RNA测序、高通量单细胞RNA-Seq 。其中,单管单细胞RNA测序,采用的是基于Smart-Seq2的扩增方法,获得全长转录本,随后Tn5转座酶进行建库测序,该扩增方法不仅可以用于转录本的定量,还可以分析结构变异和功能方面的信息。
研究流程



图1 单细胞RNA测序技术流程图
Smart-Seq扩增原理
单细胞RNA水平的研究的主要难点是单个细胞RNA含量只有pg级别,不易直接提取RNA后构建高通量文库。华大基因的单管单细胞RNA测序产品利用Smart-Seq2[2]方法,通过oligo dT 直接对mRNA进行反转录,在逆转录的过程中,利用一个叫MMLVRT(Moloney Murine Leukemia Virus Reverse Transcriptase,MMLVRT)的逆转录酶,这个酶的特性是逆转录至mRNA的5’末端后,会发挥末端转移酶的活性,在cDNA的3’端额外合成一段不依赖模板的几个碱基序列,并以逆转录体系中游离的特定碱基序列作为新的模板继续合成cDNA,最终得到了一个两端包含固定序列的全长的单链cDNA模板,随后利用此cDNA作为模板进行大量扩增。
文库构建流程
扩增得到的双链cDNA经过Tn5转座酶切打断及加接头, PCR扩增及磁珠纯化等步骤,即得到所需测序文库。构建好的文库经Agilent 2100 Bioanalyzer和Q-PCR质控合格后可进入测序环节。
测序策略
需要深入研究转录本的可变剪切、融合基因等结构功能的情况下,建议采用PE100的测序策略。仅关注转录水平的基因表达变化,建议采用SE50测序策略即可。
表1 测序策略及数据推荐
|
测序读长推荐 |
PE100 |
SE50 |
|
测序平台 |
DNBSEQ平台 |
DNBSEQ平台 |
|
测序数据量推荐 |
6 G或以上 |
20M Reads 或以上
|
信息分析内容
单细胞RNA测序能反映基因的表达差异,有助于研究基因调控网络和基因表达的异质性与随机性,结合系统生物学的方法,能应用到肿瘤研究领域。单细胞水平的基因表达调控网络分析,可监控疾病进程,持续追踪肿瘤相关基因的动态表达,对单个细胞的转录组进行测序,还可以研究胚胎不同发育阶段的组织器官图谱。以下是我们基于不同的测序读长(PE100、SE50),可进行的生物信息分析内容:
表2 标准信息分析内容
|
信息分析条款 |
信息分析内容 |
|
标准信息分析 (ref) |
1、基本数据统计① 去除接头序列、低质量序列得到reads信息,②
样品相关性,③ 表达量分布,④ RNA分类; 2、参考基因组比对; 3、mRNA鉴定; 4、mRNA定量分析; 5、mRNA差异表达分析(样本间、组间); 6、mRNA表达/差异基因聚类; 7、mRNA差异基因GO分类、富集; 8、mRNA差异基因KEGG分类、富集; 9、mRNA结构分析(SE50测序不包含此项)①
可变剪切分析,②融合基因分析(仅限人) |
|
Dr.Tom信息分析(ref) |
一)数据库注释 1、转录因子注释(AnimalTFDB/PlantTFDB); 2、GSEA分析; 3、Rfam、Pfam、Reactome、COG、EggNOG和InterPro数据库注释; 二)互作网络分析 1、靶基因分析① miRNA-mRNA靶向关系分析,② lncRNA-mRNA靶向关系分析; 2、ceRNA互作网络分析; 3、蛋白互作网络分析; 4、共表达互作网络分析; 三)特色分析 1、自定义标签和自有数据上传; 2、外部数据库信息(TCGA、ARCHS4); 3、卡方检验; 4、关键驱动基因网络图分析; 5、时间序列分析 |
|
定制化信息分析 |
可结合客户的需求,协商确定定制化信息分析内容。 |
单细胞RNA-seq对感觉传递神经元进行精确细胞亚群区分
Unbiased classification of sensory neuron types by large-scale single-cell RNA sequencing
杂志: Nature Neuroscience
影响因子: 16.095
细胞类型:小鼠腰椎神经
研究背景:
感官系统是由多种多样的细胞组成,它有很多复杂功能还没有研究清楚。文章通过对622个鼠的神经元进行测序和转录组分析,进而通过它们的明显的分子和功能的特异性对其分类:3种明显的低阈值机械性刺激感受神经元,2种本体感受神经元,6种主要的热敏神经元,痛感神经元,C型低阈值力量感受神经元,疼痛感受神经元。
文章不仅通过大规模的单细胞RNA测序,从分子水平验证了这些不同的神经元细胞类型,并发现了一些新的细胞亚型和找到这些新的细胞亚型对应的marker。例如,文章发现了皮肤炎症(例如过敏性皮炎)相关的疾病激发的疼痛感是明显的联系到特定的一种痛感神经元相关。
文章总结并发现了单细胞RNA-seq的方法可以很好的分析和研究感官反应细胞与特定类型的神经元之间的对应关系,文章的结果说明了感官细胞的多样性及其对体内感觉的复杂性等。
部分研究结果:
1. 文章对799个细胞表达量PCA聚类分析,鉴定离散的感觉神经元细胞群。鉴定结果显示神经元细胞个数为622个,非神经元细胞个数109个,68个细胞没有找到明确的定义。把这些细胞分成了五个类群,其中一个类群属于非神经元,其余四个类群是神经元。在做进一步的细胞子分类PCA聚类分析时,找到了3个新的子分类细胞群:NF1,NF2/3,NF4/5。

图1 细胞表达量PCA聚类图
图中每一个点为一个细胞,颜色反馈的是表达量的高低,根据表达谱的差异,把细胞聚成了5个类群
2. 通过GO分析,预测和区分了不同类型细胞各自对应的功能特征,同时也可以通过这些GO分析明显看出不同的神经细胞的GO功能聚类。

图2 GO功能聚类分析
3. 特异基因表达量进行功能和特征性的聚类分析,结果如图5, Ntrk2、 Ret、Calb1和Ntrk3都在NF1-NF3中表达,鉴定为低阈值的机械敏感性受体细胞类型.同时文章还发现一小群的应答和调节炎症导致的疼痛相关的细胞类型。

图3 通过对不同类群神经元进行表达量热图分析,找到亚群特征性的基因
部分内容展示:
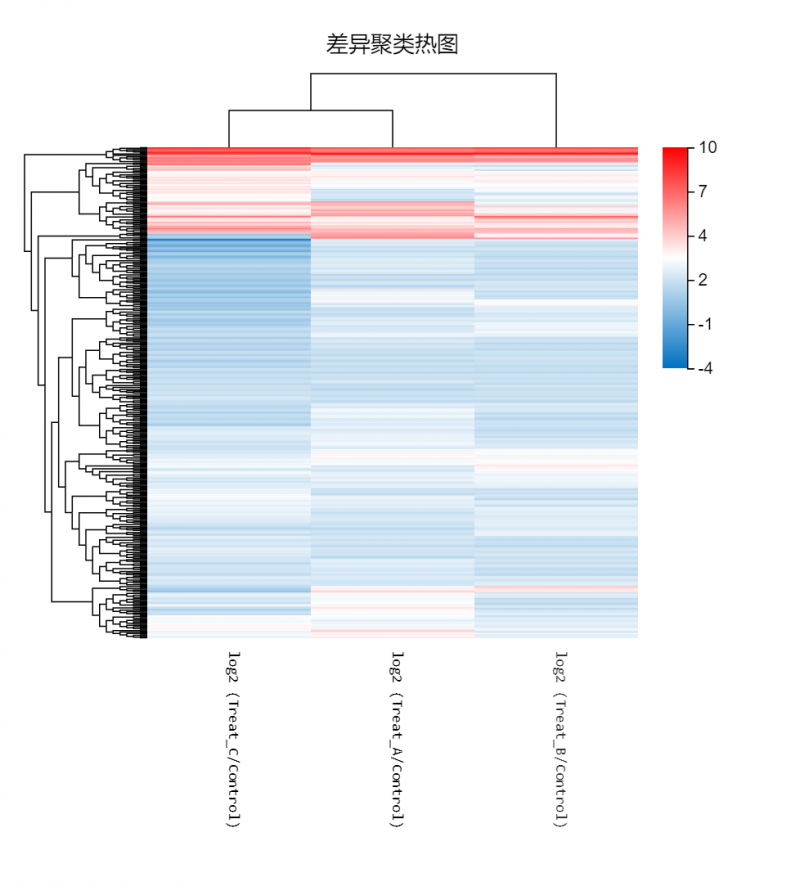
图1 差异聚类热图
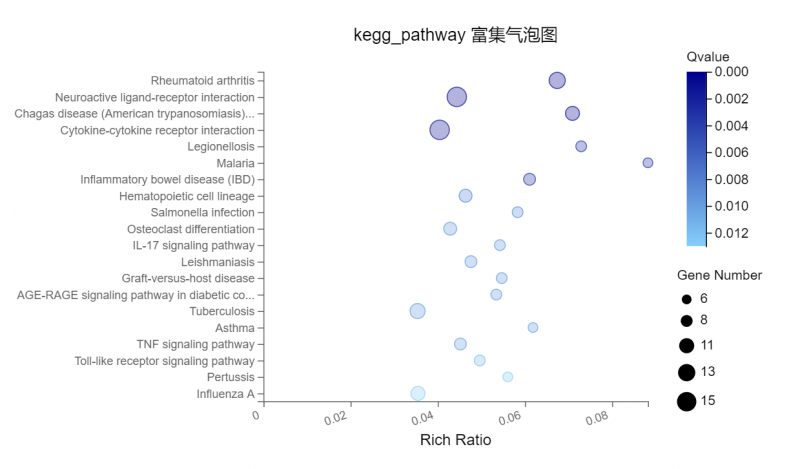
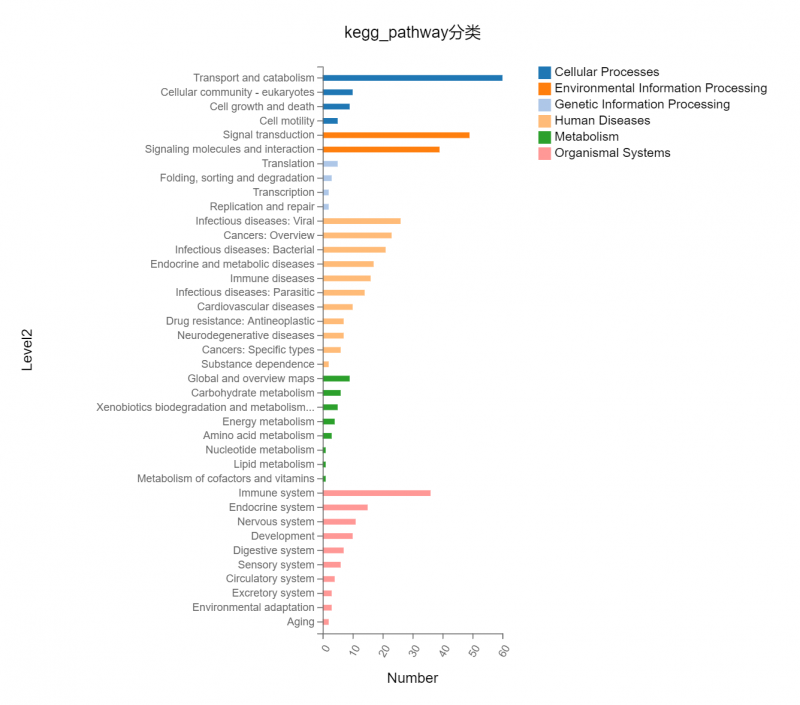
图2 功能富集或分类图

图3 蛋白互作网络
样品要求
a) 真核生物,mRNA具有Poly(A)结构。
b) 单细胞:细胞直径大于10μm,如卵母细胞、动物桑葚期至囊胚期单个细胞,细胞系分离的单细胞等。
c) 微量细胞:细胞数目在200个以下,如动物桑葚胚,囊胚。
d) 微量total RNA:一般要求RNA 28S/18S≥1,RIN≥7,浓度>50pg/uL。
表1 单细胞RNA测序送样要求
|
测序类型 样品类型 |
转录组、RNA-Seq |
|
单细胞 |
1-2个(4μL裂解液) |
|
微量细胞 |
2≤X≤200(4μL裂解液) |
|
微量总RNA |
RNA 28S/18S≥1,RIN≥7,浓度>50pg/uL |
注:表中X为细胞数目

