- 首页 > CRISPR全基因组脱靶检测
CRISPR全基因组脱靶检测
近年来CRISPR技术显著推动了基因治疗攻克血液病、肿瘤、遗传病等疾病的进程,已成为新时代科学研究领域的有力工具。然而,该技术仍存在不足之处:20 bp的识别位点特异性有限,可能造成较高的脱靶效应,最终导致严重后果。目前CRISPR系统研究重点依然在于降低脱靶率,提高特异性和准确性。
目前,脱靶的检测方法发展迅速,检测方法多种多样,也各有优劣势,华大科技基于DNBSEQ自主平台推出了CRISPR全基因组脱靶检测服务。该技术将编辑物种全基因组测序数据与参考基因组进行比对,全面检测基因编辑导致的单核苷酸突变(SNPs)以及插入缺失变异(InDels),再分析sgRNA在基因组上的同源区域从而检测可能的脱靶位点。基于此,我们助力科研用户优化CRISPR系统,从源头降低脱靶率,助力精准医学基础研究和动植物物种的快速发展。
产品优势
- 定位精确:兼具DNBSEQ™平台测序的所有优势,可以在全基因组范围内对编辑位点和脱靶位点,进行精确的定位分析;
- 无偏向性:全面分析每一个变异位点;
- 设计简单:无细胞模型限制;
- 经验丰富:大人群、大队列项目经验丰富;
- 超高通量:测序仪全面升级,可满足超高通量需求;
- 一站式全流程服务:从样本到结果,提供切实可行的全流程服务;
- 专业团队:博士级别专业团队支持。
产品应用
- 精准医学:攻克血液病、肿瘤、遗传病等疾病
- 分子育种:QTL定位、分子标记开发等
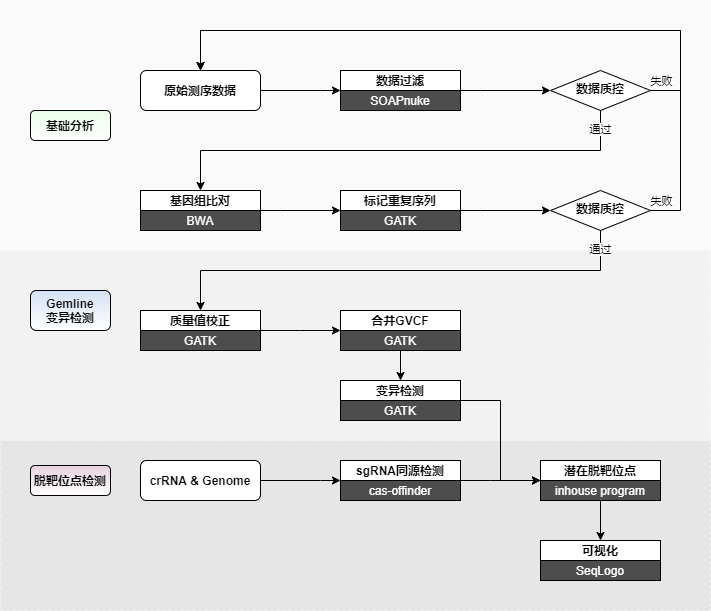
技术路线
该技术将编辑物种全基因组测序数据与参考基因组进行比对,全面检测基因编辑导致的单核苷酸突变(SNPs)以及插入缺失变异(InDels),再分析sgRNA在基因组上的同源区域从而检测可能的脱靶位点。
全基因组脱靶分析——CRISPR/Cas9编辑的原代T细胞
Genome-wide off-target analyses of CRISPR/Cas9-mediated T-cell receptor engineering in primary human T cells. Clinical & Translational Immunology. 2022
研究背景:利用T细胞抵御癌细胞是目前免疫治疗策略的一大趋势。T细胞受体(T-cell receptor, TCR)基因工程能够重定向T细胞特异性,消除同种异体反应性,同时促进了过继性T细胞转移(adoptive T-cell transfer, ACT)疗法的发展。CRISPR/Cas9 介导的DNA双链断裂使基因的敲除或敲入工程成为可能。而基于全基因组的脱靶基因检测是验证工程T细胞安全性的有效手段。
研究方案:作者基于CRISPR/Cas9技术,利用核糖核蛋白递送法敲除TCR以评估基因工程T细胞的安全性。利用全基因组测序来分析CRISPR/Cas9介导的TCR位点双链断裂是否和原代T细胞的脱靶有关。进一步通过深度靶向测序来评估全基因组测序发现的潜在低频脱靶事件。
主要结果:
1. TCR敲除:敲除TCRα链(TRAC)和TCRβ链(TRBC),TCR缺失的T细胞数量和InDel频率都显著上升,体现出表型和基因上的敲除高效性。在功能上,用超级抗原葡萄球菌肠毒素(staphylococcal enterotoxin B, SEB)刺激T细胞,敲除了TCR的T细胞干扰素IFN-γ显著降低。
2. gRNA依赖型脱靶位点预测:根据非特异性核酸酶诱导的T细胞脱靶事件来评估T细胞的安全性。Cas-OFFinder在gRNA和非靶DNA之间筛选出mismatch≤4的匹配位点,作为潜在脱靶位点。基于全基因组测序(深度约33x)检测InDel位点,包括前期软件预测的脱靶区域内的InDel。其中两个位点为预测的核酸酶诱导的低频突变位点,均未通过全基因组变异检测(GATK)过滤条件。将对照组的InDel和编辑样本的InDel进行比对,并未在两个编辑样本中检测到共有变异,故前期预测的潜在脱靶位点都未得到证实。
3. 深度靶向测序:为了进一步评估全基因组测序发现的潜在低频脱靶事件,作者对相关的基因座进行PCR扩增并进行靶向深度测序。结果显示,与对照相比,原先预测的脱靶位点并无显著性的突变。
结果展示:
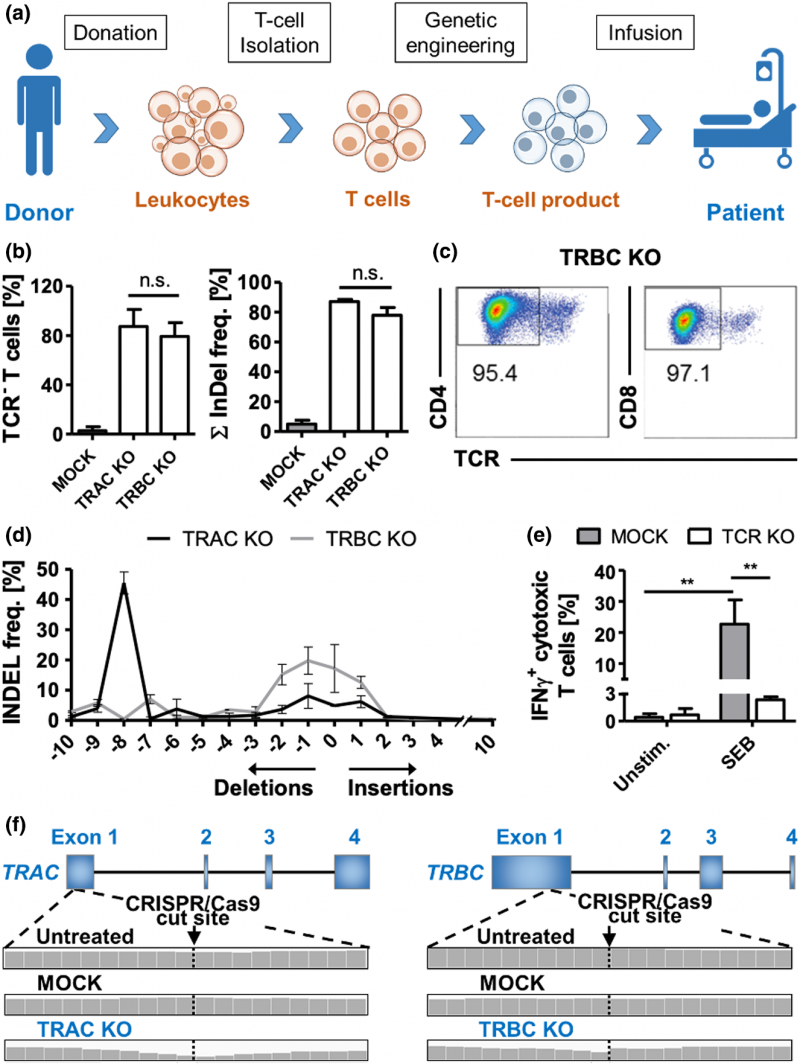
图1 原代T细胞TRAC和TRBC敲除
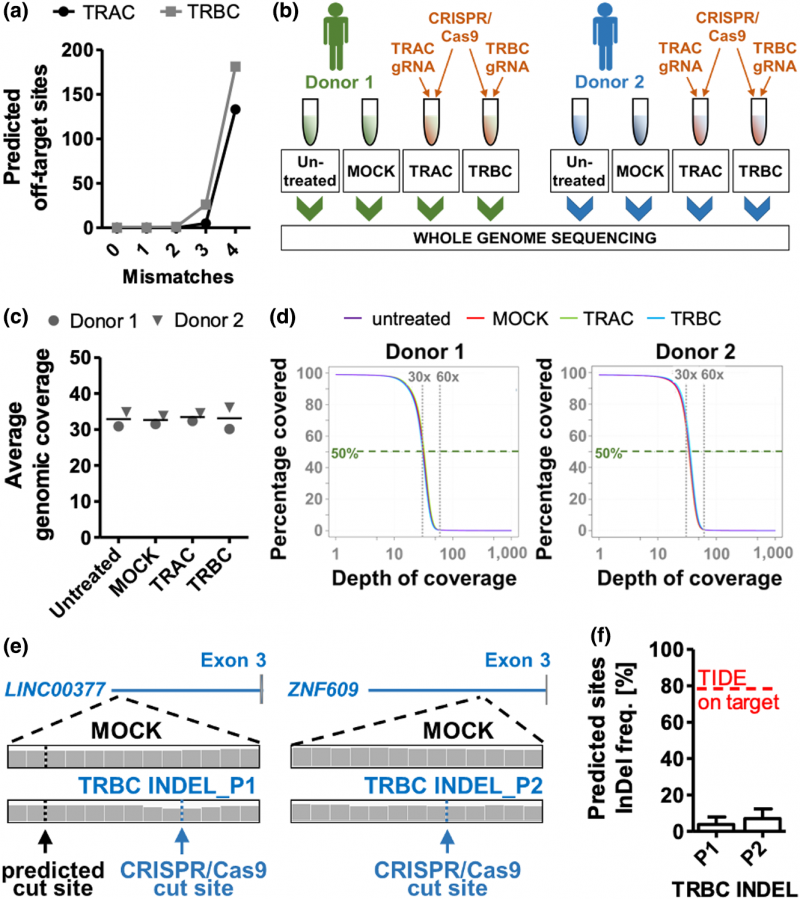
图2 CRISPR-Cas9诱导的脱靶位点预测
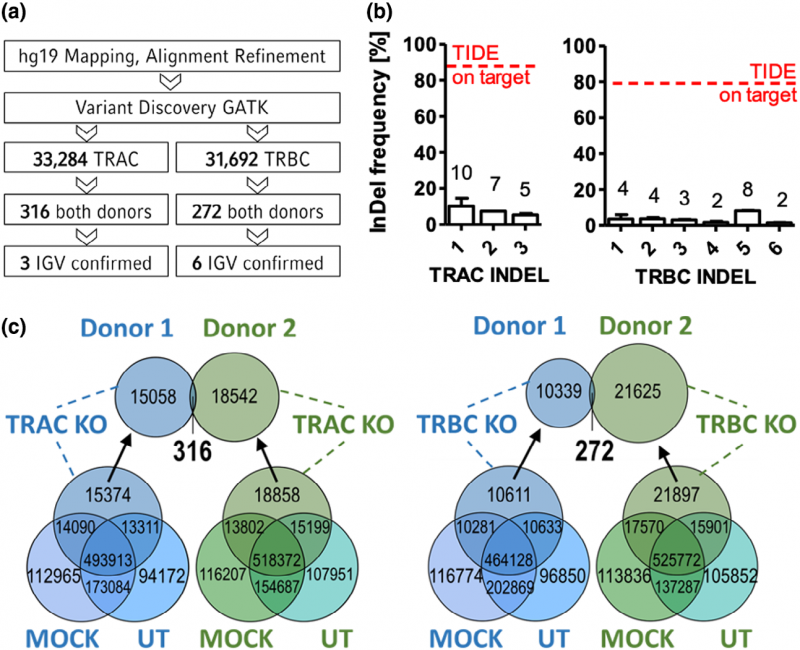
图3 全基因组脱靶基因检测
CRISPR在基因敲除、基因敲入、基因抑制和激活、多重编辑、功能基因组筛选等领域都有着卓越的成果。但CRISPR/Cas系统的广泛应用一直受到脱靶效应的阻碍,脱靶现象的存在很大程度上阻碍了CRISPR技术在生产实践中的应用,该现象可能会给宿主生物带来严重的问题。
全基因组脱靶基因检测技术可全面检测到基因编辑导致潜在的脱靶位点情况。
以下是部分分析结果展示:
1. gRNA同源区域分析
向导RNA(gRNA)是结合引导Cas9核酸酶在基因组上进行靶向性切割的RNA序列,但由于其可能对相似序列进行切割,故存在潜在的脱靶风险。运用cas-offinder软件分析sgRNA在基因组上的同源区域,脱靶位点或靶点可能发生在这些区域上。故这些同源区域上的位点将作为候选脱靶位点进入后续变异检测分析。 统计结果如表1所示。
表1 Seq1 sgRNA同源区域统计(表格为部分列展示)
|
Bulge Size |
Mismatches |
Sites |
DNAs |
|
0 |
0 |
2 |
2 |
|
0 |
5 |
8 |
8 |
|
1 |
3 |
7 |
14 |
|
1 |
4 |
76 |
140 |
|
1 |
5 |
651 |
1262 |
|
2 |
2 |
1 |
1 |
|
2 |
3 |
13 |
21 |
|
2 |
4 |
125 |
186 |
|
2 |
5 |
1356 |
2052 |
2. 变异检测
运用GATK软件检测 case 和 control 样品的 SNP 和 InDel ,筛选出同源区域的突变,并进一步过滤,结果视为潜在的脱靶位点或靶点,结果如表2所示。
表2 groupA潜在脱靶位点结果(表格为部分列展示)
| Chr | Pos | Ref | GT(case) | GT(ctrl) | DP(case) | DP(ctrl) | Change | Alt_Count | Frequence | Type | Location |
| chr1 | 206427072 | GT | GT/GT | GT/G | 30 | 41 | G>GT | 27 | 0.9 | InDel | 206427034 |
| chr3 | 5508061 | CTT | CTT/C | CTT/CT | 29 | 23 | CT>C | 10 | 0.34 | InDel | 5508095 |
| chr3 | 35451005 | CT | CT/CT | CT/C | 54 | 38 | C>CT | 54 | 1 | InDel | 35451093 |
| chr3 | 55459486 | C | C/A | A/A | 46 | 30 | A>C | 8 | 0.17 | SNP | 55459404 |
| chr3 | 62849702 | C | C/C | C/T | 20 | 32 | T>C | 20 | 1 | SNP | 62849705 |
| chr3 | 107210752 | A | A/A | A/T | 41 | 52 | T>A | 37 | 0.9 | SNP | 107210732 |
| chr3 | 130875883 | GT | GT/GT | GT/G | 15 | 14 | G>GT | 15 | 1 | InDel | 130875901 |
| chr6 | 112983632 | AT | AT/A | AT/AT | 32 | 39 | AT>A | 7 | 0.22 | InDel | 112983656 |
| chr7 | 121199013 | G | G/T | G/G | 37 | 36 | G>T | 5 | 0.14 | SNP | 121199077 |
| chr8 | 116213229 | GTT | GTT/GT | GTT/G | 20 | 20 | G>GT | 5 | 0.25 | InDel | 116213168 |
| chr9 | 107774651 | G | G/A | G/G | 29 | 37 | G>A | 8 | 0.28 | SNP | 107774669 |
| chr10 | 8090509 | TA | TA/TA | TA/T | 36 | 34 | T>TA | 36 | 1 | InDel | 8090576 |
| chr10 | 124586927 | AT | AT/A | AT/AT | 41 | 32 | AT>A | 6 | 0.15 | InDel | 124586848 |
| chr11 | 128030210 | TG | TG/TG | TG/T | 29 | 38 | T>TG | 29 | 1 | InDel | 128030258 |
| chr13 | 47520667 | AT | AT/A | AT/AT | 30 | 18 | AT>A | 9 | 0.3 | InDel | 47520556 |
| chr13 | 78715357 | TA | TA/TA | TA/T | 50 | 51 | T>TA | 50 | 1 | InDel | 78715256 |
| chr13 | 87361775 | G | G/GTATA | G/A | 24 | 18 | A>GTATA | 9 | 0.38 | InDel | 87361851 |
| chr14 | 103649698 | GTT | GTT/GT | GTT/G | 20 | 18 | G>GT | 12 | 0.6 | InDel | 103649641 |
| chr15 | 81580383 | CTT | C/CT | CTT/C | 33 | 24 | CTT>CT | 11 | 0.33 | InDel | 81580391 |
| chr16 | 66205935 | GT | GT/GT | GT/G | 24 | 24 | G>GT | 24 | 1 | InDel | 66205980 |
| chr18 | 37948858 | C | C/C | C/A | 49 | 43 | A>C | 49 | 1 | SNP | 37948879 |
| chrX | 32457061 | GA | GA/GA | GA/G | 12 | 11 | G>GA | 12 | 1 | InDel | 32457095 |
3. 可视化
Seq logo展示了潜在脱靶位点所在的sgRNA同源区域序列的碱基组成。它以图形的方式依次绘出序列比对中各个位置上出现的碱基,每个位置上碱基的累积可以反映出该位置上碱基的一致性。每个碱基对应图形字符的大小与碱基在该位置上出现的频率成正比。
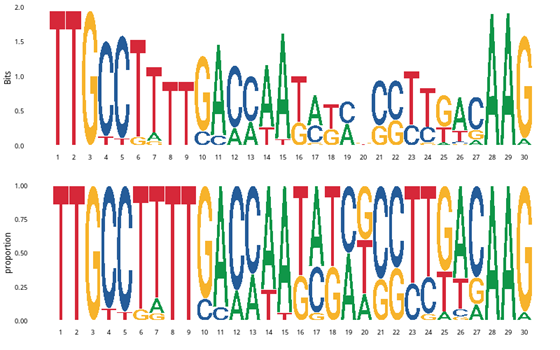
图1 潜在脱靶位点所在sgRNA同源区域碱基组成
核酸送样:
|
样品类型 |
文库类型 |
总量 |
浓度 |
完整性(胶图) |
纯度 |
|
Genomic DNA |
PCR |
≥1μg |
≥12.5ng/μL |
主峰>20kb |
无蛋白,RNA/盐离子等污染,样本无色透明不粘稠 |
|
PCR-free |
≥1μg, 推荐≥2μg |
组织送样:
|
组织类型 |
常规DNA小片段文库 |
PCR free文库 |
|
新鲜培养细胞 (细胞数) |
≥5×106cell |
≥1×107cell |
|
新鲜动物组织干重 |
≥50mg |
≥300mg |
|
新鲜植物组织干重 |
≥200mg |
≥800mg |
|
全血(哺乳动物) |
≥1mL |
≥2 mL |
|
全血(非哺乳动物) |
≥0.5mL |
≥1mL |
Q1. 怎么在微软Windows 系统中查看(打开)BAM格式的文件呢?
A1:使用Samtools工具为BAM文件创建一个索引文件 *.bai,然后用可视化软件(IGV)。
Q2. 为何我们要使用BWA-MEM呢?
A2:BWA-MEM工具用于70bp-1Mbp范围的长序列,并进行分段比对(split alignment,属于一种软剪切比对)。由于它更快、更精确,它也是最新的,所以被推荐为用于高质量的read查询、比对工具。该工具的表现评估结果可以在相关的文献中查看(Li H. 2013, arXiv)http://arxiv.org/pdf/1303.3997v2.pdf.
Q3. 该分析需要提供control样本吗?除此之外还需要提供哪些信息?
A3:需要提供control样本。除此之外还需要提供物种信息、参考基因组及gff注释文件、sgRNA序列、protospacer adjacent motif(PAM)序列以及PAM作用位置(3’端或5’端)等,详见分析方案。

