- 首页 > 长读长人全基因组重测序
长读长人全基因组重测序
人类基因组变异的完整图对于深入了解遗传特征、助力疾病精准研究非常重要。虽然短读长测序可准确地检测SNP、InDel小的变异类型,但对CNV和SV的检测灵敏度有限。近年来长读长测序已被广泛用于人类基因组研究,其能够有效解析复杂的基因组结构,包括高度重复的基因区域和基因组结构变异,为寻找疾病相关变异提供了更全面的基因组视角,其高准确性也使得它能够发现短读长测序技术可能错过的罕见变异,为精准医学基础研究提供更精确的基因变异信息。
结合华大自主研发长读长纳米孔测序技术CycloneSEQTM和短读长高通量DNBSEQTM测序技术,华大科技推出长+短WGS整体解决方案,可为科研人员提供更好基于长读长和短读长测序平台的全流程服务解决方案。
产品优势
1. 国产自主 采用国产华大自主长读长(CycloneSEQ)及短读长(DNBSEQ)测序平台,实现国产全面替代。
2. 高效精准 长读长+短读长技术融合,无偏向的覆盖基因组区域,精准解析基因组全变异。
3. 高性价比 长(15X)+短(30X),在SNP和InDel表现上均达到所有纯长读长平台30X水平,InDel表现优势显著,SV表现基本持平。
4. 全面覆盖 提供从低深度到高深度,短读长到长读长的人全基因组测序整体解决方案。
5. 经验丰富 在大规模人群队列研究领域具有深厚的项目经验,主导或参与多个国家或地区级重大项目,包括HGP 1%、千人基因组、天坛万例、瑞金ChinaMap万例、华西十万例等。
产品应用
复杂结构变异
罕见病研究
癌症研究
人群队列研究
遗传多样性研究
药物研究等
技术路线
长读长和短读长测序的下机数据,获得到高质量的reads,将reads分别比对到参考基因组上,对比对结果进行排序得到的文件联合用于后续全变异检测,最后对变异结果进行注释及统计。
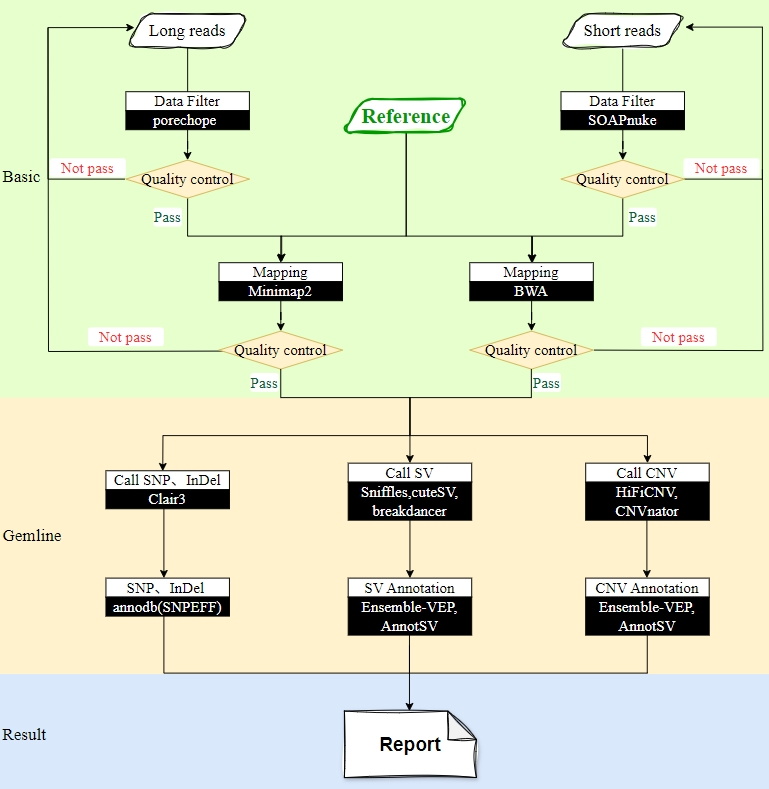
人群队列研究
Structural variation in 1,019 diverse humans based on long-read sequencing
基于长读长测序的 1,019 个不同人类的结构变异
期刊名称:Nature
发表日期:2025年7月23日
影响因子:48.5
DOI:10.1038/s41586-025-09290-7
材料:来自"千人基因组计划"26个种族的1,019人。
测序策略:ONT 平台,中位覆盖度16.9×,中位读长N50为20.3 kb。
主要成果:
(1)构建最全面的SV图谱:鉴定超过100,000个双等位SVs和300,000个多等位VNTRs,显著优于短读长测序(插入和缺失检测效率分别提高10倍和40%)。
(2)揭示SV形成机制:发现878例转座子(L1/SVA)介导的基因转导事件,其中8q21.11位点存在独特的5'转导偏好性;首次在群体水平识别1,849例倒位变异。
(3)医学应用价值:该图谱在270个临床相关位点的分型准确性显著提升,可过滤54%的候选SVs。
总结:该研究绘制了涵盖26个祖先人群中常见和罕见SV图谱。与gnomAD相比,该图谱数据中有50.9%的插入、14.5%的缺失尚未被报道,强调了长读长测序在推进SV表征方面的价值。
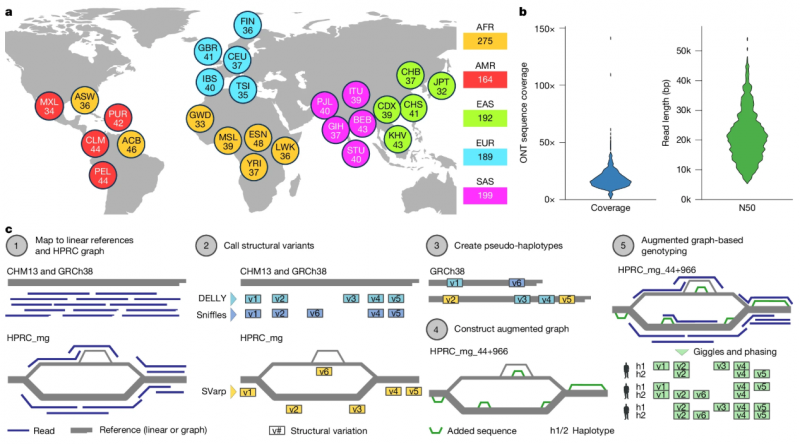
图1 取样范围和实验方法
遗传病研究
案例一:单基因疾病研究
Long read sequencing enhances pathogenic and novel variation discovery in patients with rare diseases
长读长测序增强罕见病患者的致病性和新变异的发现
期刊名称:Nature Communications
发表日期:2025年3月14日
影响因子:15.7
DOI:10.1038/s41467-025-57695-9
材料:51名高度疑似单基因疾病患者的DNA(实验组)和17例已知有基因组或甲基化畸变的DNA(对照组)。
测序策略:使用ONT平台测序(30×覆盖度,平均N50为12 kb)。
分析方法:重点筛选破坏致病基因或关键位点的变异。LRS结合自主研发的“Epimarker”工具,用于检测甲基化标记。
主要成果:
(1)该研究将罕见病诊断率提升10%,首次发现脊髓性肌萎缩症(SMA)的特异性甲基化标签。
(2)LRS可高效检测传统技术难以识别的变异:在51例未确诊患者中,LRS新诊断出5例(10%),包括2q11.2缺失综合征和SLC38A8基因突变等病例
图2 实验工具方法与样本选择
案例二:遗传病研究

- 研究疾病:遗传性血小板减少症2(THC2)
- WGS和WES测序方法无法确定该家族的发病原因。
- 使用长读长全基因组测序,确定了一个涉及配对重复倒位的大型复杂结构变体。
- 该结构变异导致致病性功能获得性WAC - ANKRD26融合转录本。
图3 研究流程与内容
罕见病研究
The implementation of genome sequencing in rare genetic diseases diagnosis: a pilot study from the Hong Kong genome project
基因组测序在罕见遗传病诊断中的应用:香港基因组计划的先导研究
期刊名称:Lancet Regional Health-western Pacific
日期:2025年1月28日
影响因子:8.1
DOI:10.1016/j.lanwpc.2025.101473
材料:来自520个具有广泛罕见遗传病谱系的家族的1264名个体,其中94%为中国人。所有样本均进行了短读长测序 (srGS),其中21例疑难样本额外接受了长读长测序 (lrGS)。
测序策略:短读长测序(平均深度39×), ONT长读长测序(30×)。
主要成果:
(1)总体诊断率达28%(146/520),其中srGS单独贡献24%,lrGS进一步提升4%。成人患者诊断率显著高于儿童(32% vs 24%),且未接受过基因检测的患者诊断率更高(37% vs 23%)。
(2)约三分之一变异为首次报道,填补了东亚人群基因组数据的空白。此外,测序技术平均缩短了15年的诊断历程,并在77%的确诊病例中改变了临床管理策略,如精准用药调整或家系筛查。
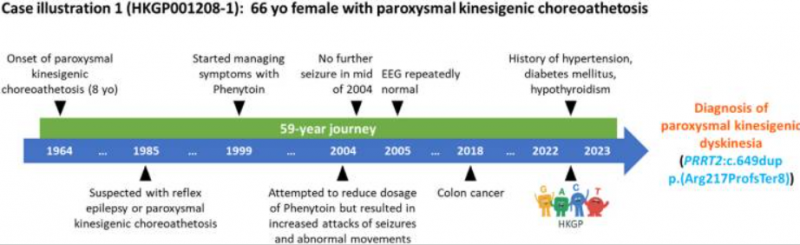
图4 诊断流程与案例说明
癌症研究
案例一:Real-time genomic characterization of pediatric acute leukemia using adaptive sampling
使用自适应采样实时表征儿童急性白血病的基因组
期刊名称:Leukemia
发表日期:2025年3月24日
影响因子:13.4
DOI:10.1038/s41375-025-02565-y
材料:57例不同基因亚型的儿童急性白血病病例的外周血或骨髓(含39例回顾性样本和18例实时诊断样本)。
测序策略:ONT 测序,平均获得了12X的全基因组覆盖度和 86X的目标基因覆盖度。
分析方法:通过自适应取样技术选择性富集59个白血病相关融合基因靶点。该方法对染色体非整倍体的检测灵敏度达96%,融合基因检出率100%,且平均9小时内即可完成。
主要成果:该技术不仅重现了传统检测(核型分析、FISH)的所有结果,还额外检出5例临床漏诊的融合事件(如DUX4::IGH),并成功识别FLT3-ITD等药物基因组变异。
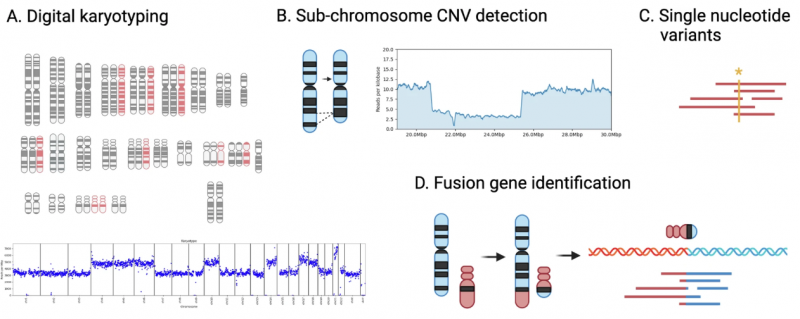
图5 实验与分析内容
案例二:肿瘤研究
Whole-genome sequencing with long reads reveals complex structure and origin of structural variation in human genetic variations and somatic mutations in cancer5.
2021年4月Genome Medicine发表了此文章。本研究对前人研究过得DNA样本进行了重新测序,证明了长读长测序技术在分析人类多态性和体细胞突变方面的优势,揭示了癌症基因组复杂结构与变异的来源,为未来的遗传学研究做出了贡献。该研究利用长读长测序技术,对ICGC(International Cancer Genome Consortium,国际癌症基因组联合会)已报道的11例日本肝癌患者进行了全基因组测序,并与ICGC测序的正常样本进行了匹配,将测序数据与参考基因组GRCH38进行比对,获得高质量SV。由于测序reads较长,大多数reads被唯一map到参考基因组,本研究中长读长检测到的SV数量是短读长的1.6倍,表明长读长测序在检测SV方面更有效。对鉴定到的肝癌患者的体细胞SV进行分析,发现长读长测序技术可以检测到大量的体细胞SV与病毒整合,与鉴定到的生殖细胞SV比较后发现,体细胞与生殖细胞中的SV存在差异。
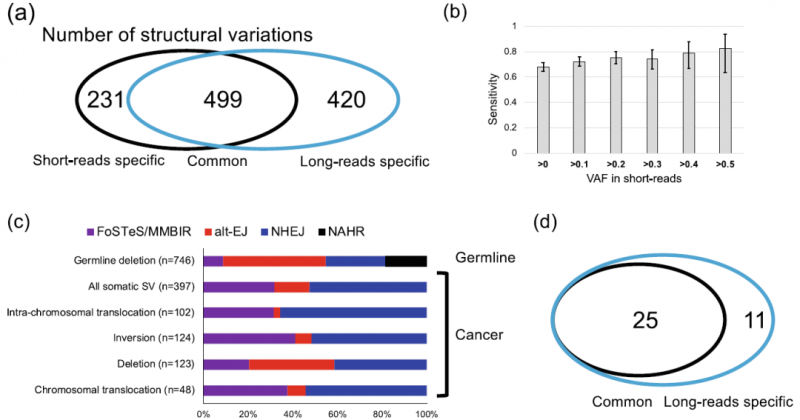
图6 肝癌中的体细胞结构变异
SNP检测准确度和灵敏度
从测试结果中看到SNP(CycloneSEQ 15X + DNBSEQ 30X)F1 Score(99.63%) 即可达到PB(30X)、ONT(30X)、CycloneSEQ(30X)水平,优于DNBSEQ(30X)。
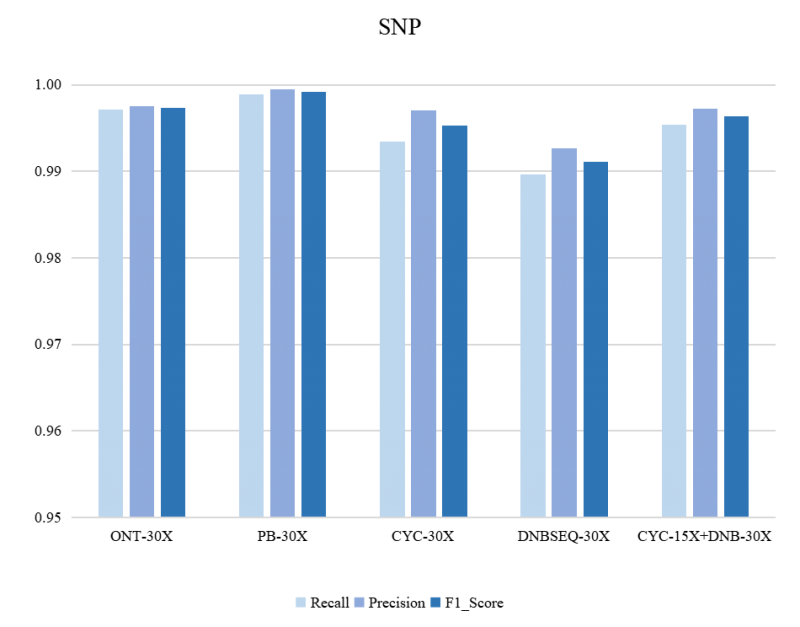
图 1 SNP检测的灵敏度和准确度
InDel检测准确度和灵敏度
从测试结果中看到InDel(CycloneSEQ 15X + DNBSEQ 30X)F1 Score (99.37%)即可达到PB(30X)水平,远超于ONT(30X)、CycloneSEQ(30X),优于DNBSEQ(30X)。
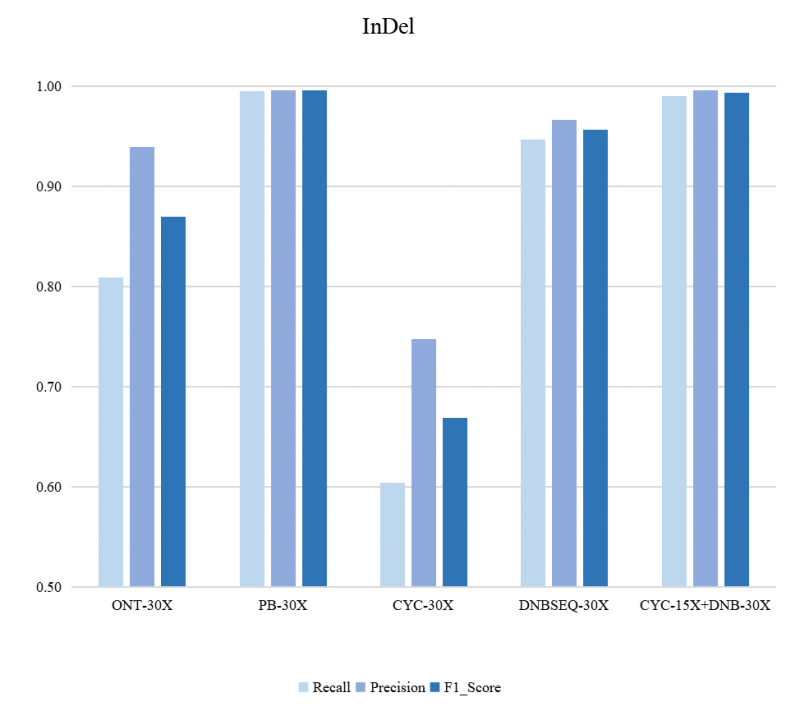
图 2 InDel检测灵敏度和准确度
SV检测准确度和灵敏度
从测试结果看到,SV(CycloneSEQ 15X + DNBSEQ 30X)F1 Score(93.83%)和PB(30X)、ONT(30X)、CycloneSEQ(30X)基本持平,远优于DNBSEQ(30X)。
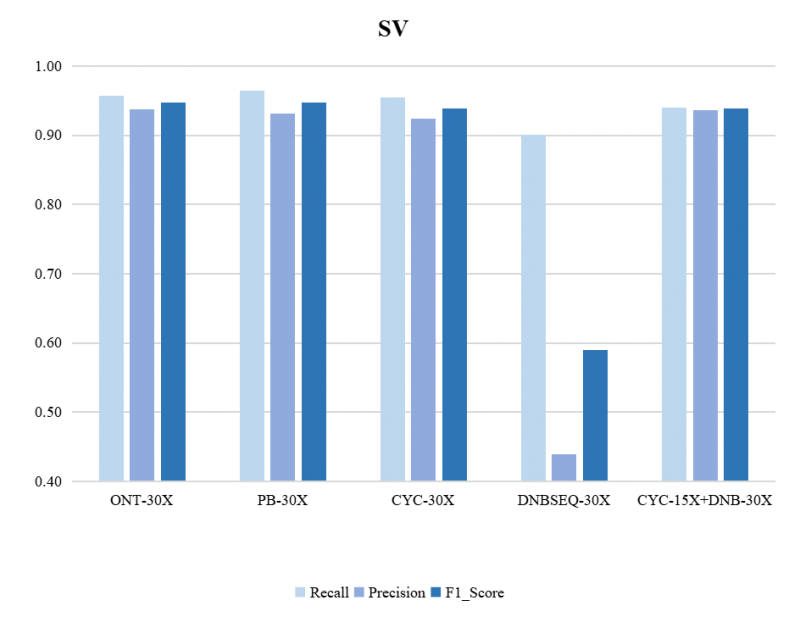
图 3 SV检测灵敏度和准确度
饱和度分析
分别截取10X,15X,20X,25X,30X,35X,40X的Cyclone数据来做深度梯度饱和度分析。从实测数据中可以看到,长读长Cyclone数据达到15X时F1值出现明显的转折,且F值达到93%以上,因此Cyclone(15X)+DNBSEQ(30X)是首选性价比较高的推荐组合方案,当Cyclone数据达到30X时,F1已经超高95%,追求更卓越质量的客户首选。
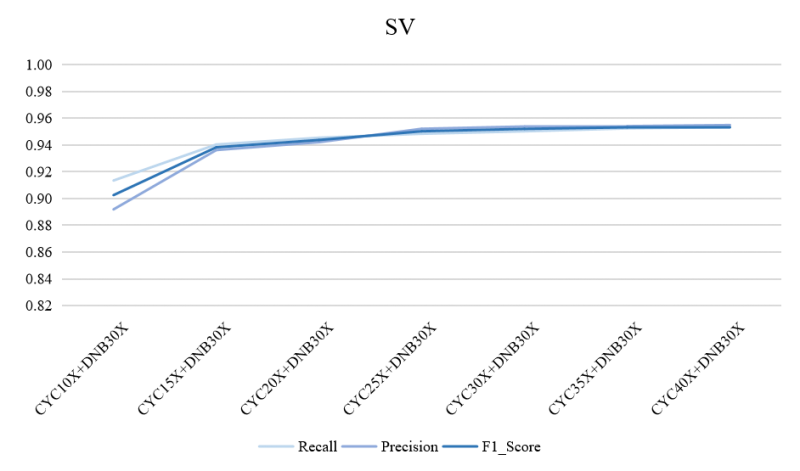
图 4 不同深度SV检测结果(长+短方案)
*上述分析结果由华大信息分析流程所得,测试结果不代表交付指标,最终解释权归深圳华大基因股份有限公司所有
DNA送样建议
样本类型 | 文库类型 | 总量 | OD值 | 样品纯度 |
Genomic DNA | PacBio HiFi CCS | m≥7μg,c≥60ng/μL | OD260/280:1.6-2.2 OD260/230:1.6-2.5 | 无RNA、蛋白质或盐离子污染; 无色透明;无粘性。 |
Genomic DNA | Cyclone normal long | m≥12μg,c≥90ng/μL | OD260/280=1.8-2.0 OD260/230=2.0-2.2 |
组织送样建议
样本类型 | 样品纯度 | |
人全血 | 新鲜血液样本(推荐) | 3-6 mL |
冷冻血液样本 | ≥6mL | |
无红细胞的血细胞(白细胞或PBMC) | 6-10mL 收集的全血 | |
细胞 | ≥5×108 | |
新鲜组织 | 组织(干重)≥1g | |
Q1: 如果自己提取,长片段DNA应该如何保存?
对于提取后的长片段gDNA样本,4°C可保存半年,-20°C可保存一年,但-20°C保存建议只进行一次冻融,反复冻融会造成样本降解,样本 DNA 不能进行震荡或剧烈混匀操作,以免长片段DNA断裂。我们不推荐您送DNA样品,因为DNA的长度直接影响了phasing的长度。另外,DNA样品在运输途中会有损伤。
Q2:为什么要用长读长检测SV?
结构变体(SV),包括缺失,插入,重复和倒位,占个体人类基因组中大多数变异的碱基对。许多研究中已经牵涉到人类健康的SV与相关表型有着直接或着间接的关系。因此,鉴定遗传变异并推断其功能影响是人类遗传学研究中最重要的问题之一。然而,因为SV易于在重复区域中出现并且内部SV结构可能出现的复杂性导致发现这些变体和基因分型变得具有挑战性。因此,需要长读长测序技术对SV结构进行新的探索,并对人类种群SV的检测进行分析,来理解与SV相关的疾病研究。
Q3: 如何选择合适的测序深度?
对于不同的研究目的和预算限制,选择合适的测序深度至关重要。一般来说,15X的长读长测序结合30X的短读长测序可以达到较好的效果。增加测序深度可以提高变异检测的灵敏度和精确度,但同时也会增加成本。因此,根据具体的研究需求来平衡测序深度与成本之间的关系是关键。

